NEJM综述:人工智能在分子医学中的应用
来源:生物世界 | 发布时间:2023-07-31
摘要:《新英格兰医学杂志》(NEJM)发表了一篇题为:Artificial Intelligence in Molecular Medicine(分子医学中的人工智能)的综述论文。
国际四大医学期刊之首的《新英格兰医学杂志》(NEJM)发表了一篇题为:Artificial Intelligence in Molecular Medicine(分子医学中的人工智能)的综述论文。

新的方法,如基因组测序和质谱技术,大大增加了科学家和医疗专业人员获取更精确诊断和增强治疗精准度所需的分子数据的数量。虽然在DNA和RNA的基因测序方面取得了最大的进展,但蛋白质和代谢物高维度测量的医疗应用也在增加。为了适应这些分子“大数据”的数量、速度和多样性,分析工具也得到了改进。机器学习的出现被证明特别有价值。在这些方法中,计算机系统使用大量数据构建预测性统计模型,并通过整合新数据进行迭代改进。深度学习是机器学习的一个强大子集,其中包括使用深度神经网络,已在图像对象识别、语音识别、自动驾驶和虚拟助理等领域具有高知名度的应用。现在,这些方法正在医学领域应用,以提供临床指导性的医疗信息。在这篇综述文章中,作者简要描述了生成高维分子数据的方法,然后重点介绍了机器学习在这些数据的临床应用中扮演的关键角色。
大规模分子数据生成
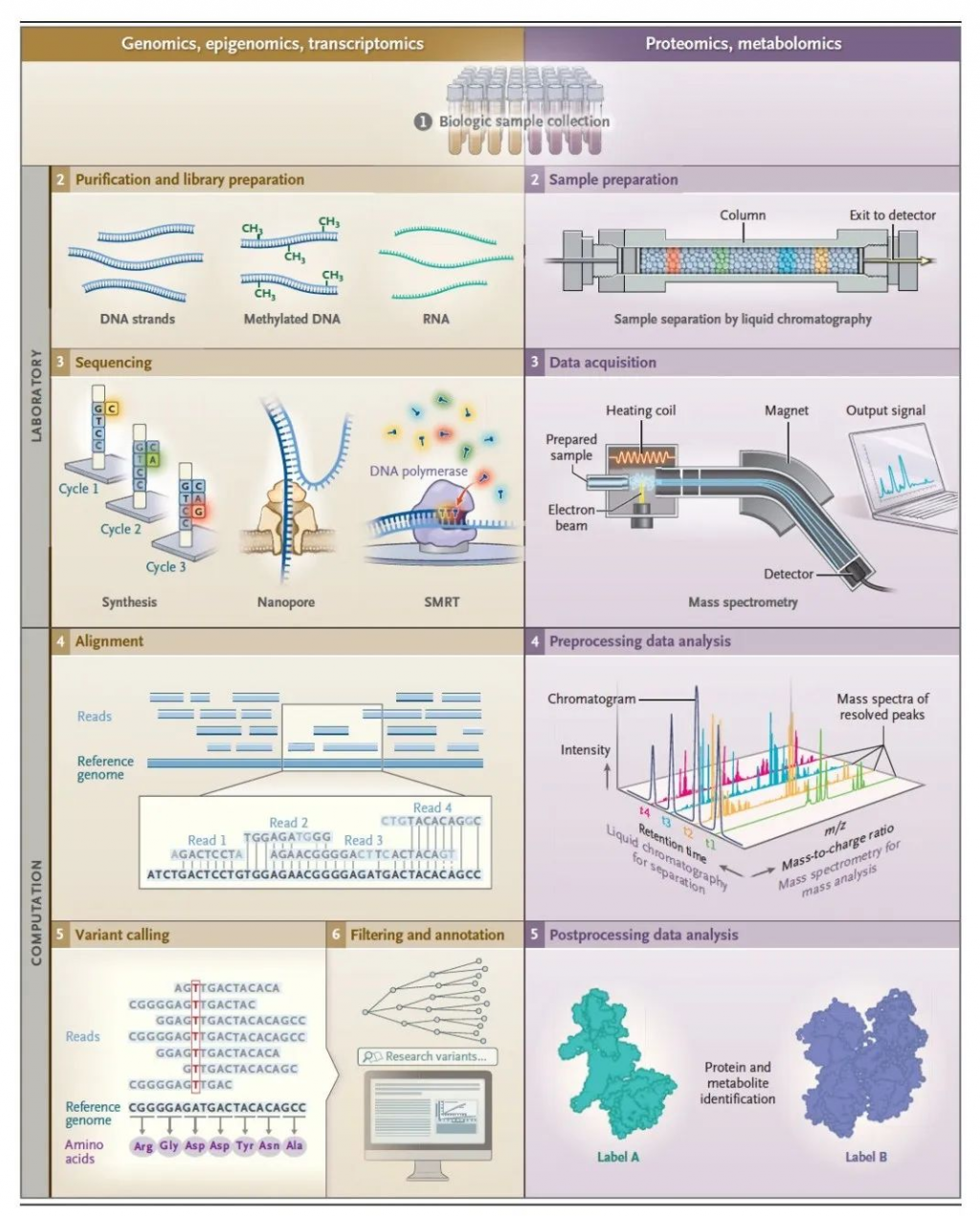
在大规模测量分子的能力方面发生了重大变化,推动了当前个体化医学的时代(图1)。几十年来,基于Sanger技术的基因测序主要集中在长度最多达几百个碱基的DNA或RNA片段上。在21世纪初,像Illumina的合成测序方法开始流行,允许同时合成和读取数百甚至数十亿个短DNA模板。更近期的方法(来自Pacific Biosciences和Oxford Nanopore)专注于连续测序长的核酸分子,具有额外的优势。而人类基因组计划花费了10年时间以数十亿美元的成本测序了一个不完整的单倍体基因组,到了2022年,一个更完整的人类基因组只需5小时的时间和几百美元的成本就能测序完成。这种基因组数据可用性的快速提高引发了对这些数据的快速处理和准确解读的需求。
基因组测序的过程会生成一个计算机文本文件,其中每一行代表一个单独“读取”的DNA或RNA分子。对于基因组测序,通常的目标是生成足够的重叠数据来覆盖基因组的每个部分40倍。某些技术可以捕获基因组的一个子集,并对其进行更多次的覆盖。这个输出的文本文件大小为100到200千兆字节(类似于现在入门级笔记本电脑的硬盘容量)。这些读取数据的长度从几百个碱基到几百万个碱基不等,通过Burrows-Wheeler变换,一种从数据压缩信息理论导出的方法,将这些读取数据映射到人类基因组计划生成的参考基因组上。然后,使用机器学习或算法方法确定正在分析的基因组与参考序列之间的差异。这样就得到了一个变异调用文件,通常有300万到400万行,几兆字节的大小。为了按照例如在患者中可能导致罕见疾病的概率对文件中的变异进行优先级排序,可以使用过滤或机器学习方法。对于RNA测序,映射后,大多数应用会侧重于基因或同工型表达的定量,而不是序列识别,将每个基因或同工型的读取计数转换为标准化的定量测量。
基因组学中的机器学习应用
机器学习在基因组学(细胞内一组基因,即基因组的研究)中的应用中,最重要的进展出现在变异检测领域:即确定分析物序列(例如来自患者的样本)与参考序列的差异位置。当将个体读取映射到参考基因组中的相应位置时,它们可以被可视化为”堆积”,其中与参考不同的碱基被突出显示(图1)。这种可视化表示有助于在基因组的复杂区域进行快速的手动审核,这一发现促使了深度学习方法用于变异识别,借鉴了计算机视觉和图像识别方面的进展。其他变异检测方法则在更狭窄的应用领域中使用机器学习,例如用于特定变异或基因组区域的技术校准误差模型。
深度神经网络是复杂的非线性函数,适用于大规模数据集。多层次的交替”神经元”权重和非线性变换将数据转化为对分类有用的抽象和低维表示。层次之间通过激活函数连接,激活函数作为输出的进一步传播的门控。在图像任务中,池化函数用于在特定区域对输入进行下采样。然后通过反向传播过程对神经元权重进行细化,并最终以对几个输出选项的置信度估计形式进行分类。卷积神经网络是一种特定形式的深度神经网络,通常用于图像识别,其特点是在图像输入上滑动滤波器的过程(图2和3)。

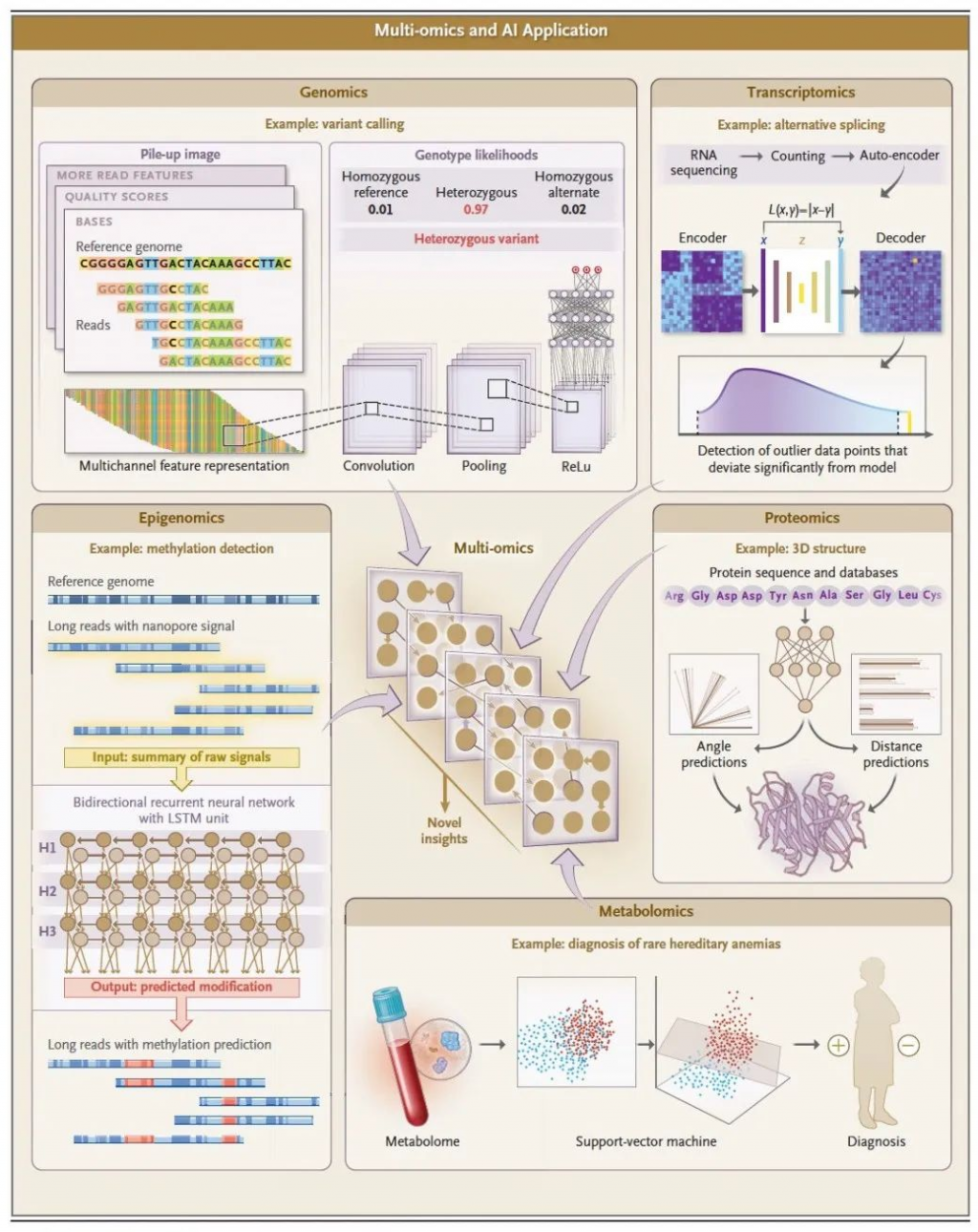
凭借神经网络的强大能力和读取更长的DNA分子的能力,可能会出现一种新的单倍型分析时代(将DNA链映射到原始亲本染色体)。单倍型分析方法通过更好地表示起源的DNA分子来提高变异检测的质量,并可以在临床管理中发挥作用,例如在复合杂合性的情况下,同一位点上两个变异体的起源父本的识别可以影响患者的护理。最近,通过将单倍型分析与针对连续数据优化的模型结合起来,然后再采用上述的卷积神经网络方法,取得了前所未有的准确性。
通过国家标准与技术研究所(National Institute of Standards and Technology)的“基因组在瓶中”联盟(Genome in a Bottle Consortium)以及美国食品药品监督管理局(FDA)的precisionFDA计划,这些进展使变异检测的改进成为可能。这两个组织共同开展了开放的“真实性挑战”比赛,使用标准化样本进行竞争。结果显示,在全基因组范围内以及染色体上编码主要组织相容性复合体等基因组中具有挑战性的区域,变异检测的准确性不断提高。机器学习(图2)在罕见疾病变异的优先级排序中也被证明非常有用。例如,一种方法使用基于逻辑回归的机器学习在一个大型的基于文献的数据集中,将表型与候选基因匹配,以帮助确定可能是门德尔遗传疾病的潜在致病基因。
转录组学的机器学习
读取转录组(生物体中所有RNA转录本的总和)被用作识别罕见疾病中的致病基因的一种额外工具。最初的努力揭示了通过将每个基因的表达谱与参考范围进行比较,识别表达异常可以指向未被怀疑的致病基因。为了获得额外的益处,这种方法后来与预测罕见变异的调节效应的贝叶斯模型相结合。在一大批患有未诊断罕见疾病的患者中,血液转录组测序识别出8%的患者的致病变异。随后,使用包括基因表达、等位基因特异表达和可选择剪接数据的分层贝叶斯模型来识别遗传驱动的转录组异常。
尽管取得了一些进展,但预测剪切位点仍然是一个具有挑战性的问题。使用一个32层深度神经网络的深度学习模型在改善罕见疾病的诊断方面显示出了希望。使用自编码器,它可以有效地学习如何将输入数据编码为压缩表示,然后解码回原始输入的表示,已经被证明可以改善从RNA测序数据中的异常剪接预测(图2)。
这些方法被应用于一个12岁女孩的案例中,该女孩出现了发育退化、震颤和癫痫。通过短读长度的基因组测序,鉴定了96个候选基因变异,但没有一个看起来与患者的病情有关。通过对患者血液进行RNA测序并应用剪切异常算法,发现了KCTD7中的一个剪切增益变异,该变异并不在最初的候选基因列表中,从而确立了进行性肌阵挛癫痫的诊断。
表观基因组学应用
表观基因组学被定义为影响基因表达的一系列修饰。虽然已知表观遗传机制在某些罕见病和常见疾病的表现中起作用,但在临床医学中,对DNA化学修饰的大规模表征才刚刚开始产生影响。长读测序方法提供了令人兴奋的机会,因为它们在核苷酸通过蛋白质纳米孔时产生信号,或者在DNA聚合酶嵌入碱基时产生信号。这些信号可以通过机器学习方法解读,不仅可以确定该位点的核苷酸,还可以确定该核苷酸的一系列化学修饰。这些方法不需要之前的标准中的亚硫酸盐转化,该转化已被证明会引起DNA断裂。由于在组织特异性转录中扮演关键角色,大部分关注集中在将甲基基团加到顺序CG二核苷酸序列中称为CpG位点的C5位点。采用各种神经网络方法,包括卷积神经网络、双向循环神经网络(图3),以及两种类型的组合,已经在甲基化检测方面达到了超过0.95的C统计量,优于以前的基准模型。
深度学习在蛋白质组学的几乎所有环节中取得了重大进展。通过对已知化学实体的光谱图模式进行训练,深度学习方法改善了候选肽段的光谱预测能力,这是基于串联质谱的蛋白质组学的关键步骤之一。利用基于卷积神经网络的工具,还能准确预测肽段的保留时间,即肽段从液相色谱柱中洗脱的时间点。除了质谱法外,深度学习应用还集中在新生肽序列和蛋白质鉴定上,采用了卷积神经网络和长短时记忆方法。此外,最近还将大型语言模型应用于蛋白质功能预测,以加速药物发现。
蛋白质的翻译后修饰,如磷酸化等过程对于蛋白质的功能、调节和降解至关重要,但其定量仍然是一个尚未解决的挑战。通过仅从蛋白质序列预测翻译后修饰位点的深度学习已经取得了成功,其中的例子包括乙酰化和泛素化等修饰。近期,通过隐藏马尔可夫模型和卷积神经网络的组合,预测蛋白质功能的能力也得到了改进。这种综合方法为360个以前未注释的人类参考蛋白质提供了功能预测,使标准蛋白质家族数据库的覆盖率提高了9%以上。
在蛋白质组学领域,基于神经网络的AlphaFold(图3)是一项备受关注的深度学习应用。它在第13届和第14届关键蛋白质结构预测评估竞赛中获得了胜利。这些是每两年举办一次的盲测竞赛,旨在评估蛋白质结构预测的进展。在第13届竞赛中,AlphaFold1为43个自由建模结构中的24个结构提供了高准确性的预测结果,大大超过了以往的方法和排名第二的方法,后者只在43个结构中的14个结构上达到了类似的准确性。在CASP14竞赛中,AlphaFold2在这一基础上取得了进一步的进展,优于许多竞争模型。
近年来,蛋白质组学在生物标志物的预测上成为临床研究的主要焦点。研究主要集中在单一标志物和多标志物的发现上。在一项研究中,利用一组配体(与蛋白质结合的寡核苷酸)实现了蛋白质定量,利用一系列机器学习模型,包括基于逻辑回归和随机森林的模型(图2和图3),对约17,000名无重大疾病的个体进行训练,预测了11个常用于预防医学的健康指标,这些个体来自五个独立的队列研究。
多组学应用
随着来自多种技术的高维数据更易获取,结合数据的计算方法变得更加重要。多组学研究的最早例子之一是对单个人进行的纵向分析,结合了基因组、转录组、蛋白质组、代谢组和自身抗体谱的数据。其他研究也采用多组学方法构建了反映健康和疾病状态的相关网络,并提出了心脑血管疾病的新生物标志物。还有一些利用深度学习的整合方法也被报道。这些方法要么在早期融合数据,将多组学数据串联起来进行单一分析,要么在后期融合数据,创建一个联合模型,将几个单一组学分析的输出结合起来。一些多组学方法在临床领域取得了成功,比如利用先前发表的神经网络等机器学习模型,将类似亮氨酸拉链转录因子1 (LZTFL1) 鉴定为COVID-19风险位点的候选效应基因。通过指出LZTFL1的表达增加可能与不良预后相关,这一发现揭示了COVID-19预防和治疗的新候选靶点。
结论
在过去的十年里,技术的进步极大地增强了我们在规模上测量基本生物过程的能力。由此产生的数据量需要越来越多针对多维生物数据集分析的机器学习方法。结果是对疾病分子轨迹的逐步详细理解,这些理解现在正在临床医学中得到应用,尤其在罕见遗传疾病的诊断和治疗方面取得了最大的进展。仍然存在挑战,包括数据质量、数据一致性和临床医生的认知。然而,随着单组学发现向多组学应用的转变,流程标准化、基准指标的扩展以及数据处理速度和准确性的提高将确保对精准医疗产生广泛影响的潜力得以实现。
原文链接: